近年来,随着高通量测序技术的快速发展,空间组学技术的关注度越来越高。
截止目前,空间组学领域已经诞生不少重要研究成果:
其中包括美国国家卫生院发布的大脑细胞普查网络计划(Brain Initiative Cell Census Network,BICCN), 项目目标旨在解析人、猴和小鼠大脑的单细胞时空动态图谱;
美国国家癌症研究所则主导了人类肿瘤图谱网络计划(The Human Tumor Atlas Network,HTAN),目的是为多种癌症各个时期的转变过程建立时空动态图谱。
一些中国高校和企业的研究团队,也已经走在空间组学科研领域的世界前沿,代表成果有国内学界绘制的首个肝癌空间图谱、以及国内业界研发的 Stereo-seq 系列技术。
与传统基因组学和转录组学研究相比,空间组学研究可以提供组织和细胞之间的空间关系信息,帮助人们深入理解生物体内复杂的细胞相互作用、信号通路、以及调控机制等生物学问题。
然而,空间组学数据处理和分析的挑战也随之而来。由于数据维度高、噪声多、复杂度大,传统的数据处理和分析方法也变得不再适用。
此外,当下至少存在数十种不同空间组学技术,按照所测的分子类别可大致划分为转录组、蛋白组、代谢组、基因组等。不同的空间组学技术也存在数据结构和数据类型上的差异,这增加了数据整合和交叉分析的难度。
对于这些快速产生的珍贵数据,科研人员面临的最直接的问题就是数据的预处理,即从原始数据处理为标准格式例如 Anndata 等。
这里的主要困难在于,不同数据类型和数据源所提供的原始数据格式并不相同,因此需要非常定制化的编程技巧。而对于特大规模数据例如 MERFISH 和 Stereo-seq 来说,更是需要大量的计算资源和时间消耗。
另一个问题在于,即使耗时耗力将某一批数据处理完毕,但在通过分析看到数据全貌之后,研究者往往会发现这个数据并非真正所需,从而导致做了无用功。

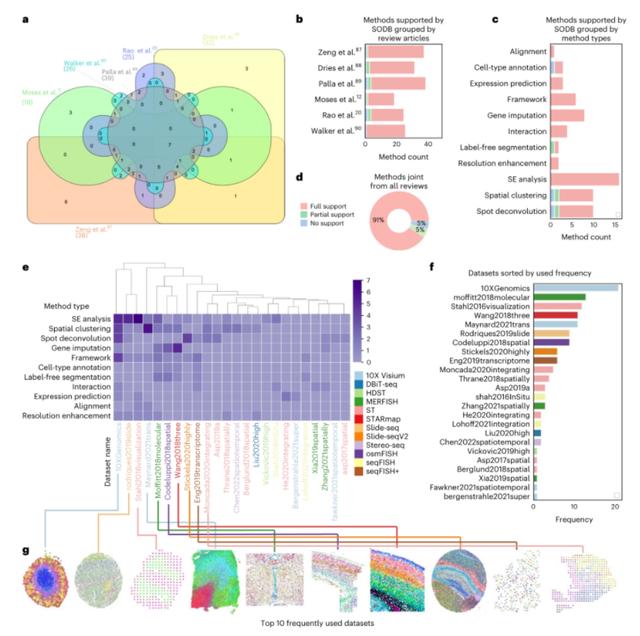
开发 SODB 数据库,数据量覆盖 5000 多万个细胞,让科研人员告别重复“造轮子”
为应对这些挑战,复旦大学类脑人工智能科学与技术研究院青年副研究员原致远,和他博士期间导师——美国德州大学达拉斯分校张奇伟教授、以及腾讯 AI Lab 首席科学家姚建华博士,开发出一种名为 SODB(Spatial Omics DataBase)的数据库,其目前数据量覆盖 5000 多万个细胞,来自 26 种不同的技术,并且这些数据均被统一地处理成标准格式,能让研究人员告别重复劳动的局面。

图 | 原致远(来源:原致远)
更重要的是,通过浏览 SODB 数据库,以及借助本次研究里的 SOView 交互式算法,可以直接“看到”空间组学数据背后的组织全貌,这样一来很容易就能判断所浏览的数据是否符合研究需求。
(来源:Nature Methods)
打个不太恰当的比喻,对于数据使用者来说,以前空间组数据像被封在集装箱中的货物,从外面看不出里面是什么货物,挑选自己所需的“商品”也非常麻烦,需要经过专业处理步骤查看集装箱里的“商品”。逛 SODB 就像“逛超市”。琳琅满目的数据就是“商品”,它们被一个个地放在“货架”上。凭借 SOView 交互式算法,用户能够看到每一个“商品”的全貌。如果满足需求,即可直接“一键购买”(下载)。而当使用本次提出的数据读取方法 pysodb 时,只需一行 Python 代码即可获取高质量数据。
概括来说,该成果的最大意义便是给空间组学领域搭建了“基础设施”,它能提供快速的数据读取和数据复用能力,满足计算生物学家在开发算法时的 benchmark 需求。
这不仅能极大地节省科研人员的宝贵时间,还可避开以往研究中的 dirty work。具体来讲,采用传统方式处理一个标准的 slide-seq 数据时,从原始数据处理到标准格式,至少需要 19 分钟的时间、以及 22GB 的峰值内存。而使用 SODB 提供的 pysodb 读取方式,仅需 7 秒的处理时间和 0.04GB 的峰值内存。
作为一个数据库,SODB 的最大意义在于能够加速空间组学领域的发展,可以帮助生物学家们更好地验证新的生物猜想,助力发现新的病理现象,降低因技术特性带来的偏见和错误性发现。
同时,SODB 还能将不同的空间数据集合在一起,使用时可以按需进行快速搜索、定位和下载多模态数据,以便充分利用已发布的数据,避免在实验上重复“造轮子”。
对于生物信息学家而言,SODB 支持多种计算方法的基准数据需求,让他们可以专注于计算模型,免去数据处理之忧。
举例来说,如果研究者只想进行简单的数据加载,那么他不仅无需提供额外的数据处理代码或将数据上传到 Figshare 等存储库中,也无需进一步研发计算方法,只需使用 pysodb 这款数据读取方法即可。
面对来自不同批次、空间技术和分子分析的异质数据,要想把它们整合起来仍然存在一定挑战。但是,之前在单细胞领域的研究已经表明,跨批次和技术整合数据具备一定可能性,并能带来更多的好处。
最近,还有研究表明将不同空间组学数据进行集成和分析,也具有较大的可行性。而这正是 SODB 可以提供的功能。
另据悉,SODB 还将促进数据的可用性,刺激空间数据整合的计算方法的发展,助力于建设具有通用坐标框架的大规模空间图谱。
当下,新兴技术在给人类带来便利性的同时,也增加了数据的复杂性。全球学界和业界都将面临越来越多的新挑战,而 SODB 可以存储、查看和分析这些数据。
同时,本次研究也有望为药物研发提供新思路。此前,空间组学技术已经被广泛用于药物研发,并在药物靶点识别、药物剂量确定和药效评估等方面展现出较大的潜力。
作为一种新工具,SODB 可以帮助研究人员深入分析细胞类型和空间关系,借此挖掘潜在的药物靶点和机制,加速药物研发的进程。
并且,SODB 还能帮助医生深入挖掘患者体内的细胞类型和空间关系,借此发现潜在的诊断标志物和治疗靶点,以便更准确地判定癌症类型和分级,从而为临床医学提供更精准的诊疗。
(来源:Nature Methods)

通过共享数据库,让更多人可以复用
担任相关论文第一作者兼通讯作者的原致远表示,本次课题要追溯到 5 年前。那时,他正在张奇伟教授的指导下开发一个名为 SEAM(spatial single nuclear metabolomics)的空间代谢组学分析方法,借此机会接触到了空间组学[1]。
在研究相关数据的过程中,他发现总得把它们挨个处理成 Anndata 格式,只有这样才能获悉每个数据的组织形态、所包含的细胞类型、细胞的分布方式等。
当手里的数据越积越多,他觉得很有必要通过开发一款在线数据库,来把这些数据共享出去,让有需要的人可以复用。“于是在和导师张奇伟教授商讨课题设计后,我跟腾讯 AI Lab 的姚建华老师和清华大学潘文韬师弟一起着手开发,并完成了数据准备工作。”原致远说。

图 | 姚建华(来源:资料图)
数据库的初版 demo 由原致远、潘文韬和赵轩(此次论文共同作者)完成,随后部署在腾讯提供的云服务器中。
几轮用户反馈过后,数据库也历经了多次迭代。于是他们开始撰写论文并投稿,在此过程中他们积极从同行研究者那里得到不少建议,例如细胞类型标注、组织识别、基因比较等。课题组不仅按照这些修改建议逐一优化,甚至还新增了可视化模块等额外功能。这些补充性开发经历了数月左右。
接着,他们继续修改论文。原致远说:“论文撰写是一个很痛苦的过程。好在在导师张奇伟教授和姚建华博士的帮助下,我们反复修改,迭代出七八个论文版本,力求以最准确客观、通俗易懂地方式让大家理解我们的工作。”

图 | 张奇伟(来源:张奇伟个人主页)
在完成项目建设和论文修缮之后,他们打算投稿给 Nature Methods,没想到期刊编辑在收到投稿邮件后一天之内就回邮件说论文即将进入同行评审环节。
“这让我们感到非常惊讶,因为通常的投稿经验得至少一周才能收到是否送审的回复。审稿过程也非常顺利,不到一个月就收到了所有审稿意见,基本都是建设性的意见,例如增加配套的 Python 分析工具。这让我们觉得前期的扎实工作非常值得,得到了专业编辑和同行专家的认可。”原致远说。
对于审稿人建议增加的 Python 分析功能,课题组非常认可其“妙处”——不仅适用于没有开发基础的生物学家,也满足了有编程基础的研发者的需求,扩大了适用人群,对于增加工作影响力大有裨益。后来才知道,这位审稿人是空间组学领域的一位奠基人,非常感谢这个建议让 SODB 增色不少。
日前,相关论文以《SODB 促进空间组学数据的全面探索》(SODB facilitates comprehensive exploration of spatial omics data)为题发在 Nature Methods 上,原致远是第一作者兼共同通讯,姚建华博士、张奇伟教授担任共同通讯作者 [2]。

图 | 相关论文(来源:Nature Methods)
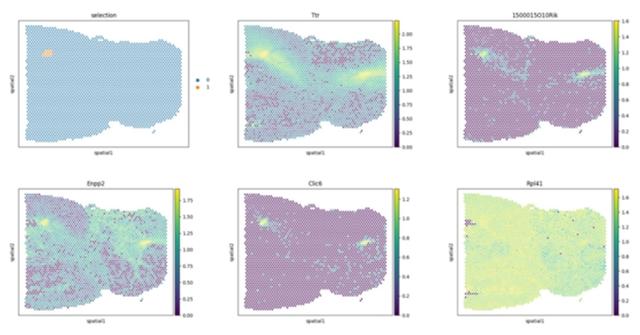
原致远表示,研究中还有不少令人兴奋的时刻。他说:“之前我们开发了一个空间组学可视化的算法 SIMS-View[1],但是只能产生静态图片。我将这个需求跟潘文韬师弟讨论后,他很快地把这个功能整合到 SODB 里,让用户可以在组织空间图谱上进行交互式探索,后来我们把它称为 SOView。”
由于 SOView 能融合丰富的基因表达信息,并能展示在组织空间中,所以它能发现一些使用传统形态学染色方法无法发现的空间域。
研究中,课题组在一个大脑数据中发现了一块很小的区域,这块区域无法被其他分析方法侦测到。而其背后存在的基因表达指纹,也被 SOView 所提供的交互式工具检测了出来。
后续,他们将继续优化 SODB 的工具性能。目前,SODB 已经可以有效处理和分析大规模的空间组学数据,但仍然存在一些性能上的瓶颈问题,因此其将进一步提高 SODB 的数据处理和分析能力。
同时,也将对数据质量加以控制。空间组学数据的质量会给分析结果带来很大影响,所以他们计划开发一套数据质量控制的工具,以用于识别和过滤低质量的数据,从而提高分析结果的准确性和可靠性。
此外,课题组也将开展应用推广和示范,即在更多的空间组学数据集上测试和验证 SODB,以证明它在实际应用中的有效性和适用性,进而开展应用示范,向目标用户介绍 SODB 的功能和应用场景,以促进其在领域内的广泛应用。
最后,该团队针对空间组学数据还开发了通用分析框架 SOTIP(Spatial Omics mulTIPle-task analysis)[3],发现了三阴性乳腺癌亚型的空间指纹,可以预测病人预后。该团队还将开展细胞类型的识别预测和功能预测。
鉴于空间组学数据中包含不同类型的细胞结构和组织结构,因此他们将进一步开发细胞类型识别和功能预测的算法,以揭示不同细胞类型的生物学功能和相互作用,从而更好地理解和阐释特定数据里的生物学信息。
参考资料:
1.Yuan, Z., Zhou, Q., Cai, L. et al. SEAM is a spatial single nuclear metabolomics method for dissecting tissue microenvironment. Nat Methods 18, 1223–1232 (2021).
2.Yuan, Z., Pan, W., Zhao, X. et al. SODB facilitates comprehensive exploration of spatial omics data. Nat Methods 20, 387–399 (2023). https://doi.org/10.1038/s41592-023-01773-7
3.Yuan, Z., Li, Y., Shi, M. et al. SOTIP is a versatile method for microenvironment modeling with spatial omics data. Nat Commun 13, 7330 (2022).
 鲁公网安备37020202000738号
鲁公网安备37020202000738号